Nous allons montrer comment se traduit cette simulation en un fichier XML que l'outil XMLlab va utiliser pour générer une simulation exécutée par Scilab. Le fichier XML sera montré sous sa forme textuelle. Seul les éléments de simulation non encore vus dans le document Prise en main XMLlab seront particulièrement détaillés.
Les lignes d'en-tête du fichier XML permettent toujours de lui
associer la DTD de XMLlab, les lignes d'en-tête de la simulation qui
suivent permettant de spécifier son titre, son auteur, sa date ainsi
que des mots-clefs qui lui sont relatifs, montrent que l'attribut
lang d'une balise de titre permet d'internationaliser
l'IHM de la simulation. C'est également le cas pour toutes les balises
de libellés destinés à être affichés.
<?xml version="1.0" encoding="ISO-8859-1"?>
<!DOCTYPE simulation PUBLIC "-//UTC//DTD XMLlab V1.4//FR"
"http://www.xmllab.org/dtd/1.4/simulation.dtd">
<simulation>
<header>
<title lang="french">Simulation de titrage de monoacides</title>
<title lang="english">Simulation of a single acid titration</title>
<author>Pauss André, Guillot Ivan, Mottelet Stéphane</author>
<date>June 2005</date>
<keywords>simulation,scilab,xml</keywords>
</header>
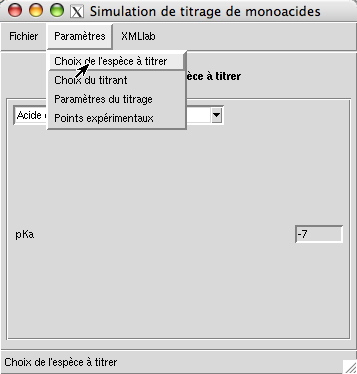
...Les paramètres de la simulation sont groupés en plusieurs sections, chaque section donnant lieu à une entrée différente du menu de la simulation, comme le montre la figure suivante :
Le choix d'une entrée provoque alors l'affichage dans la fenêtre des paramètres correspondant. Les lignes suivantes introduisent et nomment les sections de paramètres :
<parameters>
<section>
<title lang="french">Choix de l'espèce à titrer</title>
<title lang="english">Choice of the analyte</title>
...
</section>
<section>
<title lang="french">Choix du titrant</title>
<title lang="english">Choice of the titrant</title>
...
</section>
<section>
<title lang="french">Paramètres du titrage</title>
<title lang="english">Parameters of the titration</title>
...
</section>
<section>
<title lang="french">Points expérimentaux</title>
<title lang="english">Experimental data</title>
...
</section>
</parameters>Nous allons maintenant décrire en détail la déclaration des
paramètres de chaque section, dont les lignes correspondantes figurent
après les balises multilingues <title> en question
:
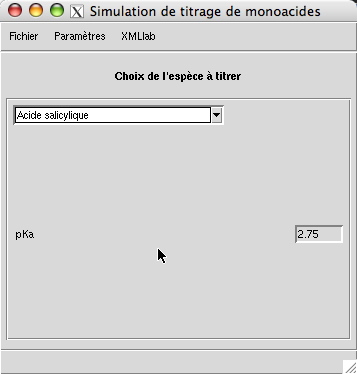
Ce choix est effectuée dans la fenêtre illustrée par la figure suivante :
Il s'agit en fait d'une banque de données contenant des paramètres scalaires (ici un seul, le pKa) pour chaque espèce à titrer. Pour chacune d'entre elles, un jeu de valeurs de paramètres est nommé. Ainsi, la figure ci-dessus présente le jeu de valeurs de paramètres .
Il est également possible de spécifier que l'ensemble des jeux
de valeurs est modifiable (cf. Section 2.2, « Balise database »
pour les détails de la balise database).
Les lignes suivantes figurent la structure de la définition de cet ensemble de jeux de valeurs :
<database>
<scalar ... label="balise_database" ...>
... définition du premier et unique paramètre
</scalar>
<record>
<name lang="french">Acide chlorhydrique</name>
... définition du premier jeu de valeurs
... pour l'ensemble des paramètres
</record>
...
...
<record>
<name lang="french">Acide salicylique</name>
... définition du dernier jeu de valeurs
... pour l'ensemble des paramètres
</record>
</database>Examinons maintenant un peu plus en détail cette structure :
Définition de l'ensemble des paramètres en spécifiant pour chacun d'eux le
labell'identifiant, son nom, le widget utilisé pour sa saisie (entrypar défaut : champ de texte), ainsi que son domaine de validité et son pas de variation, utiles par exemple dans le cas où le widget choisi est une glissière (slider). Ici, le seul paramètrepKa_analyten'est pas modifiable pour un jeu de valeurs donné :... <scalar increment="0.1" label="pKa_analyte" max="20" min="-10" state="disabled"> <name lang="french">pKa</name> <name lang="english">pKa</name> <value>4</value> </scalar> ...Définition de l'ensemble des jeux de valeurs pour ces paramètres, avec pour chacun d'eux son nom multilingue et la valeur correspondante pour chacun des paramètres définis :
... <record> <name lang="french">Acide chlorhydrique</name> <name lang="english">Hydrochloric acid</name> <scalar-value ref="pKa_analyte">-7</scalar-value> </record> <record> <name lang="french">Acide dichloroéthanoique</name> ... </record> <record> <name lang="french">Acide éthanoique</name> ... </record> <record> <name lang="french">Acide méthanoique</name> ... </record> <record> <name lang="french">Acide salicylique</name> ... </record> ...
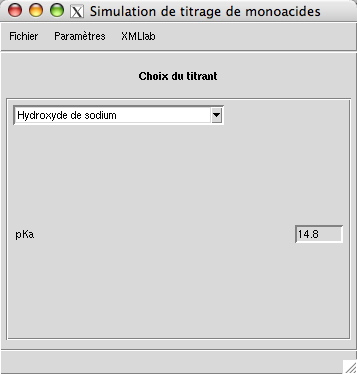
Ce choix est effectué dans la fenêtre illustrée par la figure suivante :
Il s'agit là-aussi d'une banque de données contenant un paramètre scalaire pour chaque espèce titrante.
La structure de la définition de cet ensemble de jeux de valeurs est la même que précédemment :
Définition de l'ensemble des paramètres :
<database> <scalar increment="0.1" label="pKa_titrant" max="14" min="0" state="disabled"> <name lang="french">pKa</name> <name lang="english">pKa</name> <value>14</value> </scalar>Définition de l'ensemble des jeux de valeurs pour ces paramètres :
<record> <name lang="french">Hydroxyde de sodium</name> <name lang="english">Sodium hydroxide</name> <scalar-value ref="pKa_titrant">14.8</scalar-value> </record> <record> <name lang="french">Hydroxyde d'ammonium</name> ... </record> </database>
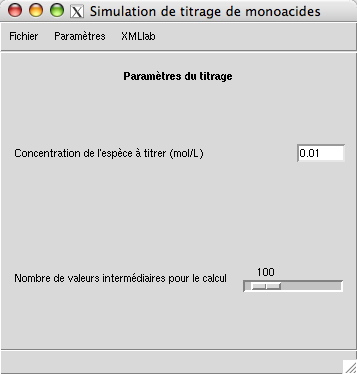
Leur saisie est effectuée dans la fenêtre illustrée par la figure suivante :
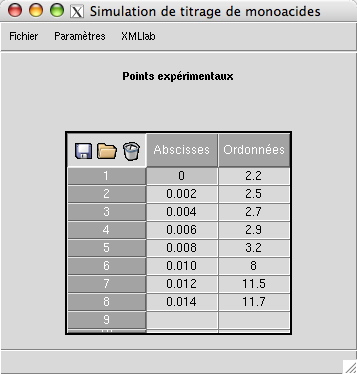
Il s'agit de saisir la concentration initiale de l'espèce à titrer et le nombre de valeurs intermédiaires utilisées pour le calcul :
<scalar increment="0.000001" label="c_analyte" max="1" min="0.000001" unit="mol / L"> <name lang="french">Concentration de l'espèce à titrer (mol/L)</name> <name lang="english">Initial concentration of the analyte (mol/L)</name> <value>0.01</value> </scalar> <scalar increment="10" label="simulation_steps" max="1000" min="10" widget="slider"> <name lang="french">Nombre de valeurs intermédiaires pour le calcul</name> <name lang="english">Number of values for the simulation</name> <value>100</value> </scalar>
Leur saisie est effectuée dans la fenêtre illustrée par la figure suivante :
Il s'agit d'un paramètre de type matriciel, les lignes
correspondant à des points expérimentaux (ici 64 au maximum), les
deux colonnes correspondant respectivement à leur abscisse et à leur
ordonnée. Elle peut être sauvegardée via l'icône ![]() dans
un fichier texte portant l'extension
dans
un fichier texte portant l'extension .dat,
chargée depuis un même fichier .dat via l'icône
![]() , ou
réinitialisée à blanc via l'icône
, ou
réinitialisée à blanc via l'icône ![]() .
.
La portion de code XML correspondant à la définition de cette matrice est la suivante :
<matrix clear="yes" cols="2" label="user_curve_mat"
load="yes" rows="64" save="yes"
striprow="yes">
<name/>
<col>
<name lang="french">Abscisses</name>
<name lang="english">X-axis</name>
<value>0</value>
</col>
<col>
<name lang="french">Ordonnées</name>
<name lang="english">Y-axis</name>
<value/>
</col>
</matrix>Cette section - qui suit la balise
</parameters> - permet de définir les équations à
résoudre, les courbes à calculer, ainsi que les domaines de valeurs
(1D ou 2D) que certaines variables doivent prendre. Ces différentes
définitions sont encadrées par les balises suivantes :
<compute> ... </compute>
En ce qui concerne notre simulation de titrage de monoacides, les éléments suivants y sont définis :
Il s'agit de la définition du domaine des valeurs discrètes
prises par la variable c_titrant au cours du titrage,
le nombre de ces valeurs étant fixé par la variable
simulation_steps (cf. § Section 2.2.3, « Paramètres du titrage ») :
<defdomain1d label="c_titrant"> <name lang="french">Concentration du titrant</name> <name lang="english">Titrant concentration</name> <interval discretization="linear" steps="simulation_steps"> <initialvalue>0</initialvalue> <finalvalue>1.5 * c_analyte</finalvalue> </interval> </defdomain1d>
Il faut préciser que la variable c_titrant peut
également être considérée comme un vecteur de toutes les valeurs
prises dans son domaine. Elle peut donc, à ce titre, intervenir dans
les calculs à réaliser comme un vecteur.
Cela consiste à utiliser une fonction implicite définissant
une application entre c_titrant et la variable
pH que l'on cherche à calculer, par l'intermédiaire
d'une équation de la forme f(c_titrant,
pH) = 0 : à chaque valeur de c_titrant
(cf. § Section 2.3.1, « Définition de la concentration du titrant »), l'équation admet une solution
unique en ph.
Les lignes suivantes figurent la structure de la définition de
la fonction implicite ph qui comprend également
:
la définition de variables de calcul intermédiaires utilisées pour calculer les valeurs prises par l'équation,
une liste de courbes en sorties, dont les abscisses et les ordonnées sont pour chacune d'elle, respectivement
c_titrantet une fonction des variables calculés.
<implicitfunction label="ph" method="dichotomic"> définition de la variable de la fonction implicite : <refdomain1d ref="c_titrant"/> <unknowns> définition de l'unique inconnue pH, dont on précise les bornes et la valeur initiale pour chaque résolution de l'équation : <unknown label="pH"> <name>pH</name> <guess>7</guess> <lowerbound>0</lowerbound> <upperbound>14</upperbound> </unknown> </unknowns> <equations> définition de notre unique équation, précédée de la définition de variables de calcul intermédiaires : <variable label="Ka_analyte">...</variable> ... <variable label="charge_titrant">...</variable> définition de l'équation dont l'annulation permet de calculer pH pour chaque valeur de c_titrant : <equation>H - (1e-14)./H - charge_analyte + charge_titrant</equation> </equations> <outputs> ... définition des courbes fonction de c_titrant </outputs> </implicitfunction>
Voici à présent le détail des variables intermédiaires de
calcul. Il est à noter que certaines variables sont des vecteurs
(comme on l'a vu pour la variable c_titrant), ce qui
permet d’optimiser le calcul des courbes de titrage par
Scilab.
A ce titre, certains opérateur Scilab
particuliers doivent être précisés (v, v1
et v2 sont des vecteurs, k un scalaire)
:
opérateur
.*:v1 .* v2 =vecteur résultant des produits deux à deux de chacun des éléments dev1etv2.k .* v = v .* k =vecteur résultant du produit de chacun des éléments devaveck.
opérateur
./:v1 ./ v2 =vecteur résultant des divisions deux à deux de chacun des éléments dev1etv2.k ./ v =vecteur résultant des divisions dekpar chacun des éléments dev.v ./ k =vecteur résultant des divisions de chacun des éléments devpark.
opérateur
.^: suit la même logique que l'opérateur./.opérateur
+et-:v + k = k + v =vecteur résultant des additions de chacun des éléments devaveck.v - k =vecteur résultant des soustractions de chacun des éléments devaveck.k - v =vecteur résultant des soustractions dekavec chacun des éléments dev.
Ces opérateurs sont donc utilisés dans notre cas, certaines variables manipulées étant des vecteurs (calcul du pH, des coefficients de partitions, des charges du titré, du titrant et du milieu), tandis que d’autres variables sont des scalaires positifs. Le tableau ci-dessous permet de distinguer les deux types de variables dans le code qui suit :
| Scalaires | Vecteurs |
|---|---|
c_analyte | |
| charge_analyte et
charge_titrant |
pKa_analyte et
pKa_titrant | charge |
| simulation_steps | c_titrant |
| |
| user_curve_mat |
<variable label="Ka_analyte">10^(-pKa_analyte)</variable> <variable label="Ka_titrant">10^(-pKa_titrant)</variable> <variable label="H">10.^(-pH)</variable> <variable label="alpha1_analyte">H./(H + Ka_analyte)</variable> <variable label="alpha0_analyte">Ka_analyte./(H + Ka_analyte)</variable> <variable label="charge_analyte">c_analyte.*alpha0_analyte</variable> <variable label="alpha1_titrant">H./(H + Ka_titrant)</variable> <variable label="alpha0_titrant">Ka_titrant./(H + Ka_titrant)</variable> <variable label="charge_titrant">c_titrant.*alpha1_titrant</variable>
Voici maintenant le détail des courbes fonction de
c_titrant qui sont définies :
<output label="alpha1"> <name>HA</name> <value>alpha1_analyte</value> </output> <output label="alpha0"> <name>A-</name> <value>alpha0_analyte</value> </output>
Il s'agit d'une pseudo courbe paramétrique, dont les abscisses
et les ordonnées sont données par les colonnes de la matrice
user_curve_mat décrite dans le § Section 2.2.4, « Points expérimentaux » :
<parametriccurve2d label="user_curve"> <name lang="french">Points expérimentaux</name> <name lang="english">Experimental data</name> <x1> <name/> <value>user_curve_mat(:,1)</value> -> première colonne de user_curve_mat </x1> <x2> <name/> <value>user_curve_mat(:,2)</value> -> deuxième colonne de user_curve_mat </x2> </parametriccurve2d>
Cette section - qui suit la balise </compute>
- permet de définir les différentes fenêtres graphiques, un ensemble
de système d'axes pour chacune d'elle (les fenêtres pouvant alors se
diviser horizontalement ou verticalement pour les contenir), ainsi que
les courbes qui doivent être respectivement affichées dans chaque
système d'axe. Ces différentes définitions sont encadrées par les
balises suivantes :
<display> ... </display>
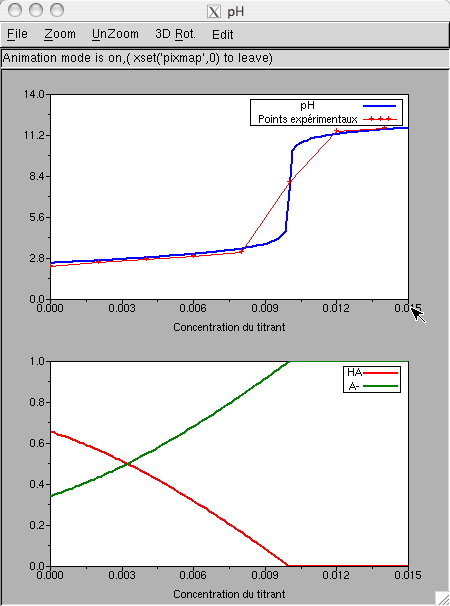
Dans notre cas, une seule fenêtre est divisée verticalement en deux systèmes d'axes cartésiens contenant les courbes suivantes :
pH calculé et pH mesuré expérimentalement, en fonction de la concentration du titrant,
coefficients de partition des différentes formes protonées de l'analyte, en fonction de la concentration du titrant : HA et A-,
Le code correspondant est le suivant, les courbes logiques définies précédemment figurant en référence dans la définition des courbes à afficher :
fonction implicite
pHet courbe des points expérimentauxuser_curve,courbe des coefficients de partition des différentes formes protonées de l'analyte :
alpha0-1,
<display>
<window splity="2">
<title lang="french">pH</title>
<title lang="english">pH</title>
<axis2d xmax="1.5*c_analyte" xmin="0" ymax="14" ymin="0">
<drawcurve2d color="blue" ref="pH" thickness="2" />
<drawcurve2d color="red" marker="plus"
ref="user_curve" thickness="1" />
</axis2d>
<axis2d xmax="1.5*c_analyte" xmin="0" ymax="1" ymin="0">
<drawcurve2d color="red" ref="alpha1" thickness="2" />
<drawcurve2d color="green" ref="alpha0" thickness="2" />
</axis2d>
</window>
</display>La sauvegarde de la session, accessible pour toute simulation par l'entrée de menu consiste à enregistrer dans un fichier l'ensemble des paramètres de la simulation, sous la forme d'un fichier de code Tcl dont l'exécution permettra ultérieurement de restaurer leurs valeurs, ceci par l'intermédiaire de l'entrée de menu . Voici une exemple d'un tel fichier pour notre simulation :
set {$}
set pKa_analyte {-7}
set {$}
set pKa_titrant {14.8}
set c_analyte {0.01}
set simulation_steps {100}
array set user_curve_mat {
39,0 39 40,0 40 5,0 5 23,0 23 5,1 0.008 5,2 2.861,0 61 44,0 44 9,0 9 27,0
27 11,0 11 48,0 48 32,0 32 15,0 15 53,0 53 36,0 36 2,0 2 19,0 19 20,0 20
2,1 0.002 2,2 2.4 57,0 57 41,0 41 6,0 6 24,0 24 6,1 0.010 6,2 8 62,0 62
45,0 45 28,0 28 12,0 12 50,0 50 49,0 49 33,0 33 16,0 16 54,0 54 37,0 37
3,0 3 21,0 21 3,1 0.004 3,2 2.6 58,0 58 42,0 42 7,0 7 25,0 25 7,1 0.012
active Abscisses 7,2 11.4 63,0 63 46,0 46 29,0 29 30,0 30 13,0 13 51,0 51
34,0 34 17,0 17 0,1 Abscisses 0,2 Ordonnées 55,0 55 38,0 38 4,0 4 22,0 22
4,1 0.006 4,2 2.7 60,0 60 59,0 59 43,0 43 8,0 8 26,0 26 8,1 0.014 10,0 10
8,2 11.7 64,0 64 47,0 47 31,0 31 14,0 14 52,0 52 35,0 35 1,0 1 18,0 18 1,1
0 1,2 2.2 56,0 56
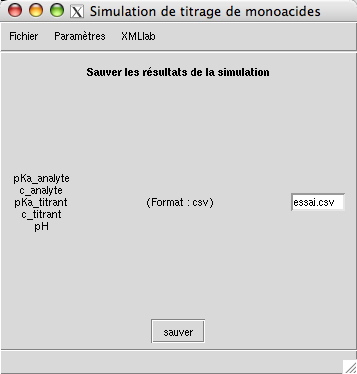
}Elle est accessible via l'entrée de menu
dès lors qu'elle a été définie via la
balise save dans le fichier XML de la simulation. Le
choix du fichier par défaut d'enregistrement et de son format, ainsi
que la liste des paramètres et résultats à y enregistrer est
déterminé par les lignes suivantes :
<save>
<file format="csv" href="essai.csv"
labels="pKa_analyte c_analyte pKa_titrant c_titrant pH" />
</save>La fenêtre suivante permet de choisir le nom du fichier qui sera enregistré ici au format CSV :
Voici l'allure d'un exemple de fichier obtenu, la balise
name de chaque paramètre ou résultat précédant sa
valeur :
"pKa : ",2.750000, "Concentration de l'espèce à titrer (mol/L) : ",0.010000, "pKa : ",14.800000, "Concentration du titrant : ",0.000000,0.000152,...,0.015000, "pH : ",2.465918,2.477587,...,11.697931,